SchNet: A Continuous-filter convolutional neural network for modeling quantum interactions
Paper Summary: SchNet: A Continuous-filter convolutional neural network for modeling quantum interactions
We propose SchNet: a neural network specifically designed to respect essential quantum chemical constraints. SchNet delivers both rotationally invariant energy prediction and rotationally equivariant force predictions.
OUTLINE:
I. Background
II. Key contributions
III. Proposed architecture of SchNet
IV. Remarks from the experiments
V. Opinion
I. Background
분자의 특성을 예측하는 것은 목적에 맞는 새로운 분자를 디자인하는데 필수적이다. 가령 약물 분자를 디자인 한다면, 분자의 독성이나 용해도 등을 예측해야 할 것이다. 하지만 탐색 가능한 분자의 공간(chemical space)은 너무나 넓어, 이러한 특성을 만족하는 분자를 실험이나 ab-initio 양자 계산을 통해 찾는 것은 시간적으로나 금전적으로 큰 부담이 된다. 따라서, 최근(참고로 이 논문은 2017년도 논문이다.)에는 딥 러닝을 이용한 분자의 특성을 예측하는 방법이 떠오르고 있다. 특히, 이번 포스팅에서는 분자의 3차원 구조를 다루는 방법에 초점을 맞춰 알아볼 것이다.
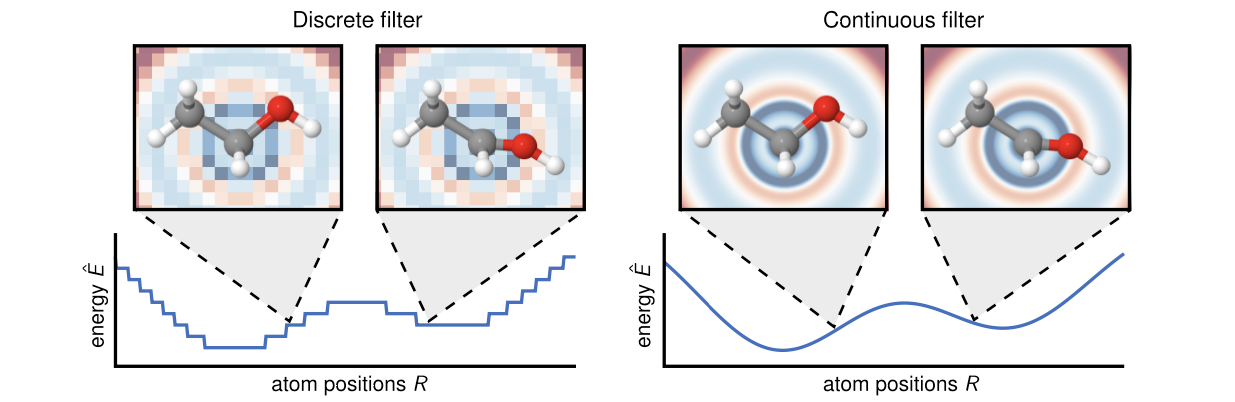
주어진 3차원 분자를 나타내는 방법(representation)에는 어떤 것들이 있을까? 제일 간단한 표현법은 아마도 3차원 격자(grid)를 이용한 방법일 것이다. 각각의 격자가 일정 간격으로 떨어진 3차원 공간상의
하지만 3D CNN을 분자에 사용하는데에는 크게 네 가지 문제가 있다. 첫 번째는, 격자 형태로 좌표를 나타내게 되면 위치의 분포가 불연속적이게 된다는 것이다. 격자의 간격을 좁히면 보다 높은 해상도로 분자를 표현할 수 있겠지만, 사용되는 표현법의 크기는
또 다른 표현법으로는 그래프(graph)를 사용할 수 있다. 그래프
II. Key contributions
본 논문에서, 거리를 one-hot vector로 표현했을 때 불연속적으로 표현되는 것을 해결하기 위해 radial basis function을 이용해 거리를 연속적으로 표현하였다. 아래 식에서
- 본 논문에서는 SchNet을 소개하며, input으로 원자의 3차원 좌표인
- ISO17이라는 새로운 벤치마크 데이터셋을 제공한다.
III. Proposed architecture of SchNet
SchNet의 구조는 아래 그림과 같다.
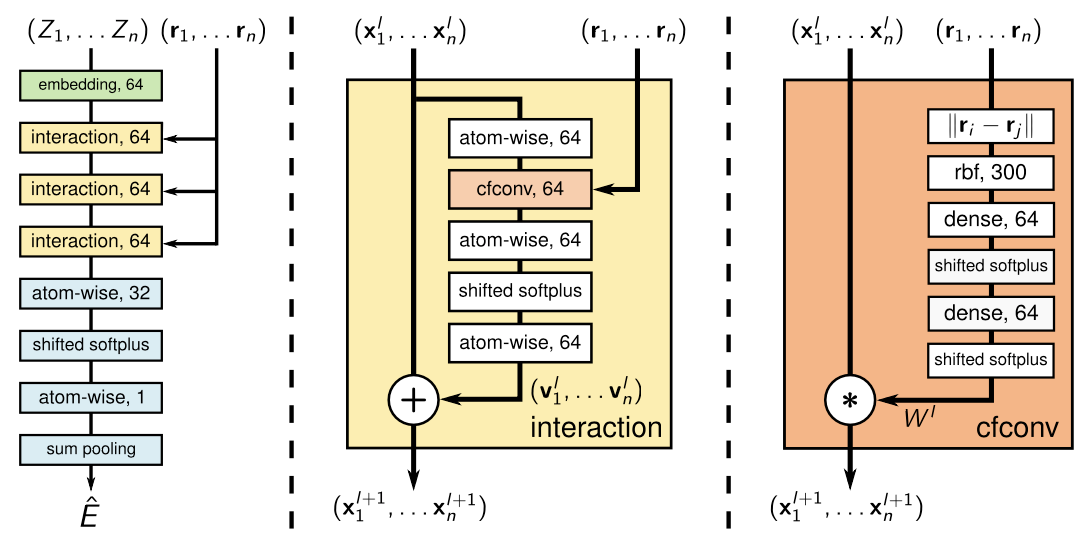
Continuous-filter Convolutional (cfconv) layer
여기서 activation function으로 shifted softplus 함수를 사용하였는데, 이때
Interaction Blocks
SchNet은 총 3개의 interaction block을 사용하였다. 기본적으로 residual connection 구조로 되어있는 interaction block은 cfconv layer를 통해 얻은
Loss Function
모델이 예측한 에너지
이를 loss function에 추가하여 실제 힘이 label로 주어졌을 때 보다 정확한 PES
IV. Remarks from the experiments
본 논문에서는 세 가지 데이터셋으로 모델을 평가하였다.
QM9
QM9은 C, N, O, F로 이루어진 약 13만개의 분자들의 equilibrium 구조를 가지고 있다. 정의에 의해

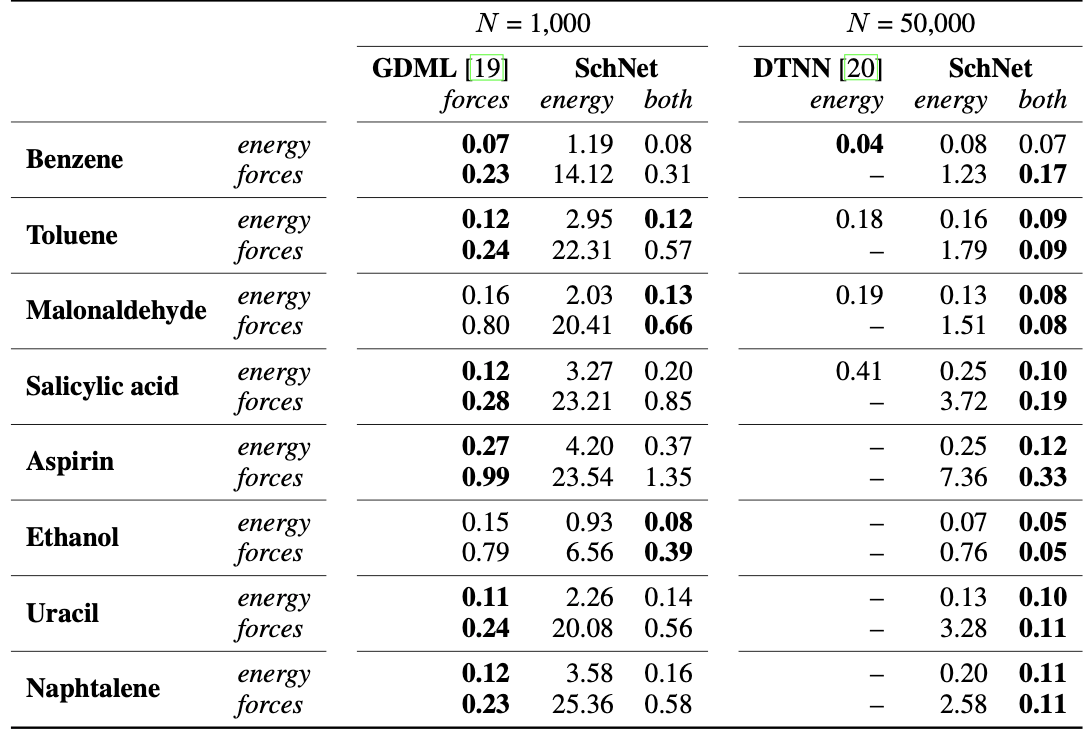
MD17
MD17은 8개의 유기 분자들의 MD trajectories를 포함하고 있다. 아래의 결과로부터 두 가지 결론을 얻을 수 있다. 먼저, energy만 가지고 학습한 SchNet보다 force를 함께 사용하였을 때의 성능이 월등히 좋아지는 것을 확인 할 수 있었다. 또한, 샘플의 수가 적을 때(
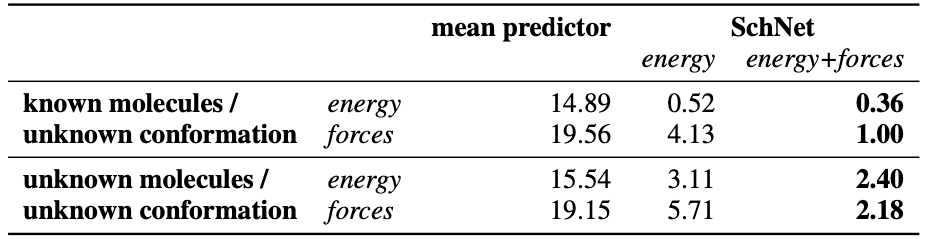
ISO17
ISO17에 대해서는 두 가지 실험을 진행하였다. 첫 번째는, train에서 사용한 분자가 test set에 있지만 구조가 다른 경우, 두 번째는 train에서 사용한 분자가 test set에 포함되어 있지 않은 경우이다. 전자는 얼마나 모델이 conformation 변화에 따른 PES를 잘 예측하는지, 후자는 얼마나 모델이 보지 못한 분자에 대해 generalize를 잘하는지 평가한다. 아래 표의 결과와 같이, energy와 force를 함께 사용하여 학습한 경우가 두 실험 모두에서 좋은 결과를 보여주었다.
5. Opinion
본 논문에서는 continuous-filter generating function을 통해 거리 정보를 연속적인 벡터로 embedding하면서 3차원 분자 구조에 대한 정보를 효과적으로 분자 물성 예측에 활용하였다. 또한, 힘-에너지 관계식을 통해 모델이 에너지를 예측하는 동시에 주어진 구조의 각 원자에 미치는 힘을 맞추게 하여 분자의 conformation에 따른 PES를 학습하도록 한 점이 인상적이었다. 또한, convolution하는 과정에서 모든 원자에 대해 동등한 비율로 summation을 하는데, 중요도를 함께 학습하여 weighted-sum을 하면 효과적일 것으로 생각된다. 다만, loss를 구하는 과정에서 derivative를 구하는 과정이 많은 계산량을 요하며, 모델이 roto-translation에 invariant하여 equivariant한 벡터 예측에는 적용하기 어렵다는 단점이 있을 것으로 생각된다.